Pro-Q Diamond Phosphoprotein Gel Stain
provides a method for selectively staining phosphoproteins in polyacrylamide gels. It is ideal for the identification of kinase targets in singnal transduction pathways and for phosphoproteomic sutdies. this proprietary fluorescent stain allows direct, in-gel detection of phosphate groups attached to tyrosine, serine, or threonine residues, without the need for antibodies or radioisotopes.
Pro-Q Diamond phosphoprotein gel stain allows detection of as little as 1-16 ng of phosphoprotein per band, depending on th ephosphorylation state of the protein. For individual phosphoproteinsm the strength of the singal correlates with the number of phosphate groups and is linear over three orders of magnitude. Pro-Q Diamond stain has excitation/emission maxima of 555/580 nm and can be detected using a visible-light scanning instrument, a visible-light transilluminator, or (with reduced sensitivity) a 300 nm transilluminator.
LLVY assay
Suc-LLVY-AMC是20S蛋白酶体,钙激活酶和其他类似糜蛋白酶蛋白酶的敏感荧光底物,荧光标记的底物通过AMC(Ex/Em = 351/430nm)产生亮蓝色荧光, 因此很容易的被DAPI滤光片所检测。蛋白酶体最通用的形式就是26s蛋白酶体,它包含一个20S的核心结构颗粒和两个19S调节调节基团。所有20S的核心结构颗粒包含四对七聚物环状结构组成两种不同的亚基,alpha亚基是结构亚基,beta亚基主要是催化亚基。在两个环状结构外每个含有七个alpha亚基主要是为了调整颗粒的停靠。Alpha亚基N端结构是可以阻挡未调节底物进入内腔的门户。内腔两个环分别包含七个beta亚基包含酶激活位点来执行蛋白水解反应。
LSM complexe
SUMMARY PARAGRAPH for LSM1
The Sm and Sm-like (Lsm) proteins are a highly conserved family of ancient origin, found in bacteria, archaea, and eukaryotes. These proteins were first characterized in humans with systemic lupus erythemathosus, where autoantibodies were found that recognize an antigen called Sm. The Sm antigen was characterized as a domain present in a group of eight small proteins (alternatively spliced products B and B’, as well as D1, D2, D3, E, F, and G) that associated with four of the five snRNAs (U1, U2, U4, and U5, but not U6) involved in splicing of nuclear pre-mRNAs (4). The S. cerevisiae genome contains 16 proteins containing an Sm, or Sm-like, domain. Seven genes encode proteins corresponding to the human Sm antigen proteins: SMB1 (Sm B), SMD1 (Sm D1), SMD2 (Sm D2), SMD3 (Sm D3), SME1 (Sm E), SMX3 (Sm F), and SMX2 (Sm G). An additional eight genes, LSM1, LSM2, LSM3, LSM4, LSM5, LSM6, LSM7, and LSM8 also encode proteins containing Sm domains and are thus named LSM (Like SM) proteins. These proteins are more closely related to individual Sm proteins than to each other. MAK31 has also been reported to contain an Sm domain (4).
Crystal structures of human Sm proteins, in pairs or with U1 snRNA, indicate that the seven core Sm proteins form a heteroheptameric ring with a small central hole (5, 6). The Sm site, the conserved uridine rich sequence found near the 3-prime ends of the U1, U2, U4, and U5 snRNAs, appears to form contacts along the inner surface of the ring complex and it is suggested that the RNA may pass through the hole (5, 6). Experiments in S. cerevisiae indicate that a similar seven-membered ring containing one copy of each of the seven Sm proteins exists in yeast (7). Similarly to the Sm proteins, the Lsm proteins also form heteroheptameric rings. In S. cerevisiae, as well as in other eukaryotes, two different Lsm ring complexes exist, containing Lsm2p-7p and either Lsm1p or Lsm8p (3).
The Lsm proteins are found in two distinct heteroheptameric complexes. Each complex contains Lsm2p, Lsm3p, Lsm4p, Lsm5p, Lsm6p, Lsm7p, and a seventh protein, either Lsm1p or Lsm8p (8). By analogy with the Sm proteins, it is thought that the Lsm proteins also form a seven-membered ring structure (9).
The complex containing Lsm8p localizes to the nucleus (3), associates with multiple snRNP complexes containing the U6 snRNA (U4/U6 snRNP, U4/U6.U5 snRNP, and free U6 snRNP) and by binding directly to the U6 snRNA, plays a role in the biogenesis and stability of the U6 snRNP and U4/U6 snRNP complexes and thus in splicing of nuclear mRNAs (1, 4, 8, 3, 9). The Lsm2-8 proteins bind specifically to the 3′-terminal U-tract of the U6 snRNA and facilitate the binding of the splicing factor Prp24p (9, 10). It is also thought that the Lsm2-8 complex plays a role in the degradation of nuclear RNA substrates by targeting them for decapping (11).
The complex containing Lsm1-7p is cytoplasmic and mutations in LSM1-7 all cause defects in mRNA degradation (2). Lsm1-7p are found in association with the Pat1p decapping enzyme and Xrn1p exoribonucleases; thus the Lsm1-7 complex is thought to be involved in mRNA degradation via the decapping step (8, 3, 12).
There may be yet another Lsm complex, containing at least six members (Lsm2-7p), as these six proteins, but not Lsm1p or Lsm8p, have been found in association with the pre-RNase P RNA (3, 4) and with the box H/ACA snoRNA snR5 (13).
Because it has been observed that cells depleted of any of the essential Lsm proteins (Lsm2-5p or Lsm8p), are defective in processing of tRNA (14), rRNA (15), and snoRNA (16), it has been suggested that the Lsm proteins may have the additional role of directly processing these classes of RNAs. However, it has also been observed that these phenotypes are alleviated by overexpression of the U6 snRNA, so it may be that these other RNA processing defects are downstream effects of lowering U6 levels and thus inhibiting splicing (13).
20091028
Wilson et al. BBA-Review mRNA surveillance pathways
Pathways of normal mRNA degradation
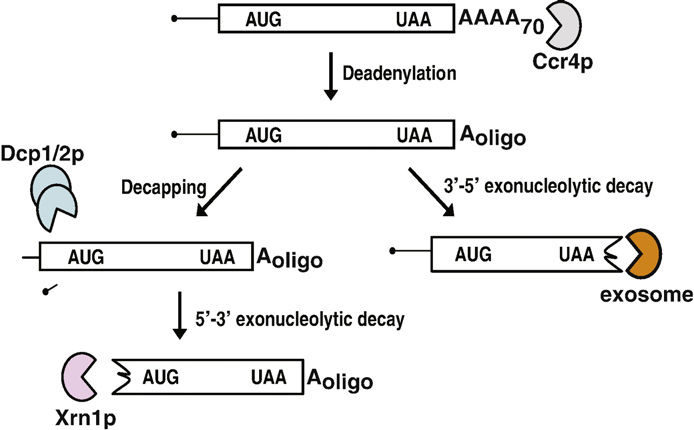
2 major cytoplasmic degradation pathways for normal mRNAs. All of the enzymes required for these two pathways are conserved in other eukaryotes, suggesting that the pathways of mRNA decay are also conserved. Messenger RNA degradation is initiated by gradual removal of the poly(A) tail, a process which is normally carried out by the 3′ exonuclease Ccr4, but also by Pan2, at a slower rate. In the absence of both Ccr4 and Pan2, poly(A) tails are stable, suggesting that there are no other enzymes that can substitute for this function. Removal of the poly(A) tail triggers two mRNA degradation pathways. In the first pathway, the 5′ cap is removed by the Dcp1/Dcp2 complex. Decapping of the mRNa triggers degradation of the transcript from the 5′ end by Xrn1, a 5′ exoribonuclease. In the second pathway, the body of the transcript is degraded from the 3′ end by a multi-subunit 3′ exoribonuclease termed the exosome. The exosome has both nuclear and cytoplasmic functions, both requiring additional factors. For example, Ski2, Ski3, Ski7 and Ski8 are required for all cytoplasmic exosome functions.
NSD-Nonstop mRNA decay
which selectively degrades transcripts that lack all in-frame termination codons. The translation ribosome translates to the end of the poly(A) tail of a nonstop mRNA and stalls. The stalled ribosome is hypothesized to be recognized by Ski7, possibly because of the absence of a codon in the A-site of the ribosome. Recognition by Ski7 recruits the exosome to the nonstop mRNA, resulting in degradation of the transcript.
Several lines of evidence support the current model. First, it is likely that the translating ribosome stalls at the end of a nonstop transcript. Nonstop mRNAs remain physically associated with ribosomes when used in in vitro translation reactions, while ribosomes dissocieate from mRNAs that contain a stop codon. Stalled ribosome-mRNA complexes have been useful tools in studying the sorting of nascent protiens, and are surprisingly stable, as they can be purified by sucrose gradient centrifugation or gel filtration. Thus, unlike DNA or RNA polymerases, ribosomes do not simply dissociate when they reach the end of a temlate, which implies that a specific facgtor (perhaps Ski7) is necessary for disassembly of the ribosome. Similarly, the bacterial ribosome needs trans-acting factors to dissociate when it reaches the end of a nonstop mRNA (i.e. tmRNA and SmpB)
Second, nonstop mRNA decay requires active translation. Nonstop mRNAs are stabilized in wild type yeast cells treated with a translational inhibitor, and in mutant cells depleted of charged tRNAs. More importantly, translation of the nonstop mRNA itself is needed: nonstop mRNA decay is prevented when a stop codon is inserted close to the poly(A) tail, and when a stable structure in the 5’UTR prevents its translation.
Third, a nonstop PGK1pG reporter mRNA was stabilized in yeast strains lacking cytoplasmic exosome function, suggesting that the exosome degrades nonstop mRNA. In contrast, mutations inactivating the decapping enzyme or the 5′ to 3′ exoribonuclease, xrn1p, have large effects on the degradation of normal mRNAs, but do not detectably affect the stability of the nonstop PGK1pG mRNA.
Fourth, based on sequence similarity, Ski7 is a likely candidate for recognizing the stalled ribosome at the end of a nonstop mRNA. The C-terminal domain of Ski7 is homologous to translation factors eEF1A and eRF3. Because eEF1A interacts with the ribosome when the A-site contains a sense codon and eRF3 interacts with the ribosome when the A-site contains a stop codon, it is proposed that the homologous domain of Ski7 interacts with the ribosome when the A-site is empty. This hypothesis is supported by the observation that deletion of the C-terminal domain of Ski7 inactivates the nonstop mRNA decay pathway without affecting other exosome functions.
Fifth, the N-terminal domain of Ski7 inteacts with the exosome, and with additional exosome cofactors. This domain is required for both nonstop mRNA deacay and other cytoplasmic exosome functions. Importantly, a point mutation in the exosome that disrupts its interaction with Ski7 also blocks nonstop mRNA decay, confirming that the interaction between Ski7 and the exosome is functioally important.
The exosome degrades the poly(A) tail on nonstop mRNAs is surprising since the exosome is incapable of degrading the poly(A) tail on normal mRNAs, as shown by a complete absence of deadenylation in a ccr4Δpan2Δ double mutant. The dependence on translation and the independence of a distinct deadenylation phase appear to be conserved aspects of nonstop mRNA decay.
2 questions à Résoudre:
1. Whether the translation rate of a nonstop mRNA is the same as for a control mRNA?
2. The fate of the protein produced from a nonstop mRNA is also unclear.
Reports from 2 groups indicate that nonstop proteins may be targeted for proteolysis by the proteasome. Inhibitors of the proteasome increased the levels of the protein encoded by nonstop mRNAs, and a screen to identify trans-acting factors in nonstop mRNA decay uncovered three mutations that inactivate the proteasome. Interestingly, the effects of the proteasome and Ski7 are additive, suggesting that recognition of the stalled ribosome by Ski7 is not required for the rapid degradation of the encoded protein.
Premature polyadenylation generates nonstop mRNAs
1.2% of random yeast cDNA clones and 0.7% of human cDNAs were prematurely polyadenylated. Premature polyadenylation can also serve as a method of gene-specific regulation. In S. cerevisiae, 0.8% of all open reading frames contain a cryptic plyadenylation site upstream of the normal termination codon. Cryptic sites could be favored over the normal poly(A) site under various conditions to down-regulate the expression of the normal mRNA.This implies that premature polyadenylation, coupled with nonstop mRNA decay, could serve to down-regulated many genes.
NGD-No Go mRNA decay
Pseudoknots, rare codons, or stem-loops within an open reading frame cause the translating ribosome to stall. Transcripts with stalled ribosomes are degraded by the no-go mRNA surveillance pathways. A stalled ribosome within the coding region of the no-go transcript is thought to recruit Dom34 and Hbs1. Dom34 and Hbs1 are homologous to eRF1 and eRF3, respectively, suggesting that they may function to recognize stalled ribosome. Interaction of Dom34 and Hbs1 with the stalled riboosme is thought to trigger endonculeolytic cleavage of the no-go mRNA. The resulting 5″ cleavage product is degraded by the cytoplasmic exosome, while the 3′ cleavage product is degraded by the 5′ exoribonuclease, Xnr1. Consistent with this model, an xrn1Δ strain accumulates the expected 3′ cleavage product, but deleting DOM34 from this strain prevents this accumulation. Likewise, a strin lacking cytoplasmic exosome activity accumulates the expected 5′ cleavage product, and deletion DOM34 from this strin prevents this accumulation. Similarly, mutants in HBS1 show drastically reduced levels of no-go mRNA decay intermediates, but hbs1Δ is not as effective as dom34Δ, implying that Hbs1 may not play as great a role as Dom34 in this pathway. Consistent with this observation, recent structrural work suggests that Dom34 is responsbile for endonucleolytic cleavage of no-go mRNA.
Ribosomes can also stalled because of aberrancies within the ribosome. Mutations that are prefentially degraded through the nonfunctional ribosomes that are preferentially degraded through the nonfunctional ribosome decay pathway (NRD). Thus, in the cas of no-go decay, a stalled ribosome leads to mRNA decay, while in NRD, a stalled ribosome leads to rRNA decay. Yet in other cases of ribosomal stalling, yeast mRNAs are stabilized ( e.g. treatment with cycloheximide or depletion of charged tRNA in a cca1-1 stain). Thus, an important question yet to be addressed is how the cellular machinery distinguishes between whether the mRNA or rRNA in a stalled complex is defective, or whether in both cases both mRNA and rRNA are degraded.
An interesting aspect of the relationship between nonstop and no-go decay is that Ski7 and Hbs1 are a pair of duplicated genes in yeast, whereas most other eukaryotes have only one homolog. The single Ski7/Hbs1 homolog from the related yeast Saccharomyces kluyveri could complement the growth phenotypes of an HBS1 mutant and a SKI7 mutant in S. cerevisiae. In contrast, the human homolog of Hbs1/Ski7 is unable to complement either an HBS1 mutant or a SKI7 mutant in S. c. the most likely explanation is that human eRFS is nonfunctional when expressed in yeast, leaving the question whether eRFS can function in nonstop and/or no-go decay unanswered. The evolutionary history of Hbs1p and Ski7 suggests the possibility taht in most eukaryotes, the single Ski7/Hbs1 protein recognizes stalled ribosomes within the coding region and at the end of an mRNA. This also suggests taht in other eukaryotes, nonstop and no-go mRNA decay may be a mixture of endonucleolytic decay and exosome-mediated decay.
20091025
Fernandez et al. 2004 MBC-(Lsm2-Lsm7 complex)
In yeast, Lsm2-Lsm8 complex binds and stabilizes the 3′ end of the spliceosomal U6 snRNA, the Lsm2-8 proteins likely form a heteroheptameric ring;
whereas the Lsm1-Lsm7 complex functions in mRNA decay, Capped mRNA degradation intermediates accumulate in yeast containing mutations in these proteins, indicating that Lsm1-7 complex functions in mRNA decapping. As 3′-shortened mRNAs accumulate in mutant strains, the Lsm1-7 complex may protects 3′ ends of deadelylated mRNAs from nculeases. Immunofluorescence experiments reveal that Lsm1 is mostly cytoplasmic, whereas Lsm7, a component of both complexes, is nuclear and cytoplasmic. Within the cytoplasm, Lsm1 localizes to structures called p-bodies, whch represent sites of mRNA degradation. Thus, the Lsm2-8 complex may be nuclear, consistent with its role as a U6 snRNP component, whereas the Lsm1-7 complex appears largely cytoplasmic.
Lsm2-Lsm7 associates with snR5, a box H/ACA snoRNA that functions to guide site-specific pseudouridylation of rRNA. snR5, this RNA is a member of the box H/ACA class of snoRNAs that fucntion in pseudouridylation of rRNA. Thre are >20 box H/ACA snoRNAs in yeast, all of which are bound by four core proteins: Gar1, Nhp2, Nop10, and the pseudouridine synthase Cbf5. Approximately half the snR5 RNA in cells i sbound by Lsm proteins. The Lsm complex bound to snR5 is distinct from Lsm2-8 and Lsm1-7 complexes, because neither Lsm1 nor Lsm8 are associated with the snoRNA. Experiments in which binding of Lsm proteins to snR5 was reconstituted in vitro reveal that the 3′ end of snR5 is required for Lsm protein recognition. We demonstrate that components of the Lsm2-7 complex are prsent in nucleoli. Interestingly, biochemical fractionation and immunoprecipitation experiments suggest that at least some of the Lsm2-7/snR5 complex is distinct from the fraction of the snR5 bound by Gar1 and Nhp2. Consistent with a separate complex, Lsm proteins are not required for the function of snR5 in pseudouridylation.
Spiller et al. 2007 Jounal of cell science-(Lsm2-8 complex)
Nuclear accumulation of Lsm8 requires Kap95. Production of recombinant human LSM proteins in bacteria, followed by injection of thse prteins into HeLa cells, showed that the pre-assembled LSM2-8 complex localized to the nculeus, whereas LSM injected by itself accumulated in the cytoplasm. These results suggest that LSM8 nuclear import involves an unidentified nuclear-import signal that is only present when LSM8 interacts with other LSM2-8 subunits.
The three paralogues yeast Lsm proteins, Lsm8, Lsm2, Lsm4 also contain basic C-termini, they might form a nculear-localization signal in a similar fashion. Mutations in yeast Lsm8 are suppressed by overexpression of LSM2 or LSM4.
Under normal physiological conditions, competition between Lsm1 and Lsm8 might provide a link between RNA processing events in the nucleus and mRNA degradation in the cytoplasm.
Lsm proteins are actively imported through the nuclear pore
nup49-313 (a nuclear-pore mutant taht affects protein import), strain ts
In contrast to the effect of the nup49-313 mutation on Lsm protein localization, the xpo1-1 nuclear-export mutation showed no effect on the localization of Lsm1, Lsm7 or Lsm8. Thus, it seems that the cytoplasmic localization of Lsm1 is not a consequence of nuclear exclusion by continual active export from the nculeus, at least not through this export pathway.
Nuclear localization of Lsm7 requires other Lsm proteins (not Lsm1)
lsm1Δ and lsm6Δ heat sensitive
Depletion of Lsm2 or Lsm4 disrupts Lsm8 localization
Lsm8 by itself does not accumulate in the nucleus, because loss of either of its proposed partners in the lsm2-8 ring results in its delocalization. However, it seems that a complete Lsm2-8 complex is not essential, because lack of Lsm6 has no effect, which might be expected because Lsm6 is a non-essential protein.
Lsm7 and Lsm8 delocalization is not caused by defective splicing
depletion of Lsm2-8 proteins leads to decreased levels of U6 snRNA and an accumulation of per-mRNA.
Lsm8 truncations affect its nuclear localization, Lsm8 needs to interact with other Lsm proteins for its nculear accumulation
Over-production of Lsm1 or Lsm8 has opposing effects on Lsm7 localization
lsm8-1 mutation is synthetic lethal with deletion of LHP1 (which encodes the yeast homolog of La, another U6 RNA-binding protein), and that the requirement for Lhp1 in an lsm8-1 strain can be suppressed by low-copy overexpressiono f LSM2.
The proposed existence of an Lsm2-7 complex that associates with snR5 in thenucleolus is in apparecontradiction with our finding of Lsm8 requirement for nuclear localization. It seems possible that the full Lsm2-8 complex might interact with snR5, but that the Lsm8 epitope tag might be masked in the snR5 RNP.
Pannone et al. 2001 Genetics-(lsm2-8 complex)
A model of Lsm2 and Lsm4 contact Lsm8 in the Lsm2-8 ring.
Deletion of LSM5, LSM6, or LSM7, but not LSM1, are synthetically lethal with deletions of LHP1. Lhp1 acts redundantly with the assembled Lsm2-8 complex to stabilize newly synthesized U6 RNA.
2009-09-02
1. Actively transcribed rRNA genes in S. cerevisiae are organized in a specialized chromatin associated with the high-mobility group protein Hmo1 and are largely devoid of histone molecules.
Katharina Merz,1 Maria Hondele,2 Hannah Goetze, Katharina Gmelch, Ulrike Stoeckl, and
Joachim Griesenbeck3 (2008 Genes Dev)
2009-07-29
Ampicillin inhibits cell wall biosynthesis. Cleavage of the B-lactam ring by B-lactamase (bla gene) is the mode of resistence.
Chloramphenicol inhibits translation of the 50S ribosomal subunit at the peptidyltransferase step (elongation inhibition). Acetylation by chloramphenicol acetylgtransferase (cat gne) is the mode of resistence.
Kanamycin binds to 70S ribosomal subunit, inhibits translocation and elicits miscoding. Aminoglycoside modifying enzymes (acetyltransferase, phophotransferase and nucleotidyltransferase) alter the antibiotic, preventing its interaction with ribosomes and inactivation it.
Streptomycin inhibits protein synthesis. It binds to the S12 protein of the 30S ribosomal subunit, causing misreading or inhibiting initiation. Mutation in the rpsL (gene for S12 ribosomal protein) prevents binding of streptomycin to the ribosome. Aminoglycoside phosphotransferase also inactiviates this antibiotic.
Tetracycline inhibits protein synthesis (elongation) by preventing binding of aminoacyl-tRNA to the ribosome. Loss of cell wall permeability is the mode of resistance.
2009-07-21
10# Cole et al. 2009 Mol Cell-(nonfunctional rRNA decay)
Quality control of eukaryotic transcripts begins in the nucleus. Nuclear RNA surveillance has been most extensively studied in S. cerevisiae and relies heavily on the exosome, the cell’s major 3′-5′ exonuclease. The nuclear and cytoplasmic exosomes share ten core subuntis, one of which, Rrp44p, is responsbile for its exonucleolytic activity. However, nuclear and cytoplasmic exosomes differ by the presence of the exoribonuclease Rrp6p and nucleic acid binding protein Rrp47p in the nucleus versus the GTPase Ski7p in the cytoplasm. Within the nucleus, the exosome has been shown to degrade aberrant pre-mRNAs, pre-tRNAs, pre-rRNAs and pre-snoRNAs. Prior to their decay, many nuclear exosome substrates undergo polyadenylation by the Trf4/5o/Air/Mtr4p polyadenylation (TRAMP) complex, which stimulates decay via exosome recruitment. Decay of such polyadenylated RNAs is thought to occur within a region of the nucleolus known as the No-body. Come polyadenylated pre-RNAs are also thought to be decayed by the nculear 5′-3′ exonuclease Rat1p, although where this occurs within the nucleus is unknown.
In the cytoplasm, several pathways have been described for mRNA quality control. all of these pathways are translation dependent, initiating when a ribosome stalls during translation in a context that impedes efficient elongation or termination. Nonstop mRNA decay (NSD) eliminates mRNAs lacking any in frame stop codon, such as trancated or prematurely polyadenylated transcripts. This pathway is dependent on Ski7p, which recruits the cytoplasmic exosome. A second quality control system, nonsense-mediated mRNA decay (NMD), eliminates mRNAs containing a stop codon in a poor context for translation termination, often a nonsense or premature termination codon. Following recruitment of th eUpf proteins (Upf1p, Upf2p and Upf3p) to the stalled translation complex, the mRNA is decapped and then degraded by the major cytoplasmic 5′-3′ exoribonculease Xrn1p. Nonsense transcripts are also subject to 3′-5′ degradation by the cytoplasmic exosome via interaction between Upf1p and Ski7p. Finnaly, no-go mRNA decay (NGD) eliminates mRNAs containing a structural barrier within the open reading frame that induces ribosome stalling. Such stalling stimulates endonucleolytic cleavage of the mRNA immediately upstream of the structural barrier, followed by Xrn1p- and Ski7p- mediated decay of the 3′ and 5′ halves, respectively. Within the cytoplasm, both general mRNA turnover and NMD are thought to occur in discrete structures known as processing or P-bodies. P-bodies are gegions in eukaryotic cells that contain translationally repressed mRNPs and proteins involved in mRNA decay. These proteins include the decapping cimplex Dcp1p/Dcp2p, decapping activator Dhh1p, and exoribonuclease Xrn1p. Where in the cytoplasm NSD and NGD occur, however, has not been previously examined.
Mature tRNAs undergo rapid tRNa decay (RTD) by a process involving Rat1p and Xrn1p. rRNAs containg deleterious mutations in either the peptidyl tansferase center of 25S rRNA or the decoding site of 18S rRNA are subject to a late-acting quality control system dubbed nonfunctional rRNA decay (NRD).
Cycloheximide is an antibiotic that inhibits the peptidyl transferase activity of ribosomes engaged in translation elongation, effectively locking them onto mRNA via an interaction with the large subunit.
18S NRD is a cytoplasmic porcess. P-bodies also function in aberrant rRNA elimination.
25S NRD appears unrelated to any of the knwon ranslation-dependent mRNA decay pathways. Unlike NMD, NSD, NGD and 18S NRD, 25S NRD still occurs in the presence of translation elongation inhibitors. Further, 25S NRD substrates accumulate around the nuclear envelope as opposed to the dispersed cytoplasmic localization observed for 18S NRD substrates in WT cells. Finnaly, 25S NRD is independent of all mRNA decay factors tested, with teh exception of the core exosome exonculease Rrp44p. 25S NRD must recruit the exosme independent of Ski7p.
2009-04-28 thèse de Anne-Laure Finoux
Transcription:
l’initiation de la transcription:
La synthèse d’un ARN pré-messager a lieu au cours de l’étape de transcription qui est très finement régulée. chez les eucaryotes, différentes ARN polyùérases existent et chacune d’entre elles est responsable de la synthèse d’ARN spécifiques. Ainsi, l’ARN polymérase I est responsable de la production de la majorité des ARN ribosomaux (ARNr), l’ARN polyméase III de la synthèse des ARN de transfert (ARNt), de l’ARNr 5S et d’autres petits ARN, et l’ARN polymérase II transcrit les ARNm, certains snRNA (small nuclear RNA), snoRNA (small nucleolar RNA) et les micro-ARN. La polymérase II représente le centre d’une machinerie complexes auquel viennent s’ajouter six facteurs de transcription généraux (TFII-A, -B, -D, -E, -F, -H) responsables de la reconnaissance du promoteur et du déroulement de l’ADN et un large complexe applelé médiateur impliqué dans la régulation de la transcription. Le coeur de l’ARN polymérase est constitué de douze sous-unités protéiques capable de synthétiser des molécules d’ARN et possédant une activité de correction des erreurs. Les promoteurs sont les lieux d’assemblage du complexe de préinitiation et sont constitués de régions constantes et de régions régulatrice. L’élément de base d’un promoteur est constitué d’une séquence TATA et/ou d’une séquence d’initiation (Inr) contenant le site de démarrage de la trascription et eput également contenir un troisième élément appelé DPE (downstream promoter element) situé en aval du site d’initiation. Les élém »nts régulateurs sont quant à eux spécifiques des gènes et localisés en amont du coeur du promoteur. Is incluelent des séquences activatrices (UAS: upsteam activation sequences) et inhibitrices (URS: upstream repression sequences) qui servent de sites de liaison pour les activateurs et inhibiteurs de la transcription. Chez la levure S. cerevisiae, les éléments TATA sont situés 40 à 120 nucléotides en amont du site d’initiation de la trascription et ont pour séquence consensus la séquence TATAAA. Ils représentent le site de liaison de la protéine TBP (TATA binding protein) qui est essentielle pour une initiation efficace et pour l’assemblage du complexe de transcription. Notons qu’il existe des promoteurs dépourvus de séquence TATA et dans ce cas, la transcription des gènes correspondants demeure dépendante de la fixation de TBP.
La régulation de la transcription s’effectue essentiellement au niveau de l’étape d’initiation. Il existe deux grands types de régulation transcriptionnelle chez les eucaryotes. D’une part, de façon générale, la structure de la chromatine ne permet pas l’accès de la polymérase au promoteur en raison de son organisation en nculéosomes. Ceux-ci représentent ainsi des répresseurs généreux de l’expression génique. Ce degré de régulation est corrélé aux différentes modifications post-traducctionnelles des queues des histones (méthylation, acétylation, phosphorylation, ubiquitination) qui permettent le recrutement d’enzymes de remodelage de la chromatine. Notons qu’il a récemment été mis en évidence que ces modifications existent également dans la région codante des gènes, régulant ainsi les différentes étapes de l’élongation de la transcription. D’autre part, la régulation transcriptionnelle requiert l’intégration d’informations provenant de facterus de transcriptions spécifiques liés aux séquences activatrices (UAS) régulées situées en amont du démarrage de la trascription. Cette intégration est réalisée par le médiateur qui représente un large complexe protéiques pontant l’ARN polymérase II et les nombreures protéines impliquées dans l’activation et la répression de la trascription. Ainsi, les facteurs de trascription liés à ces séquences interagissent directement avec le médiateur mais pas avec la polymérase. La polymérase II, les facteurs de transcription généraux et le médiateur forment un large complexe de 2.5 MDa qui s’assemble à chaque promoteru spécifique de la polyméarse II avant l’initiation.
Très tôt après l’initiation, et de façon concomitante à la transcription a lieu l’agout en 5′ d’une structure particulière importante pour l’ARN néoformé: la coiffe.
Couplage de la transcription et de l’ajout de la coiffe:
Au cours de l’étape de transcription, l’ARNm subit un certain nombre de modification. Les premières étapes de ces maturations sont physiquement et fonctionnellement couplées à la transcription. Le premier évènement est l’ajout en 5′ d’une structure particulière grâce à la machinerie d’addition de la coiffe. Ce complexe intervient très tôt au cours de la synthèse de l’ARN puisque celui-ci ne fait alors qu’environ 22 à 25 nucléotides. Le recrutement de cette machinerieest réalisé par le domaine C-terminal de la grande sous-unité de l’ARN polymérase II -CTD) qui peut être considéré en partie comme une plate-forme assurant la liaison de nombreux facterus de maturation des ARN mais nous verrons également par la suite son implication directe dans le couplage de la transcription avec d’autres étapes de la maturation. Ce domaine comprend plusieurs répétititons d’un heptapeptide de séquence consensus YSPTSPS. Il peut adopter plusierus conformations de fçon dépendante de son état de phophorylation. Ainsi, la sérine 5 du CTD est phophorylée lorsque la transcription est initiée, puis la sérine 2 l’est au cours de l’étape d’élongation.
La réction d’ajout de la coiffe peut $etre divisée en trois étapes majeurs. D’une part une première enzyme élimine un phosphate de l’extrémité 5′ de l’ARNm. La réaction suivante correspond à l’ajout d’un résidu guanosine par une guanylytransférase via son recrutement par le domaine CTD phophorylé au niveau de la sérine 5. L’enzyme est ensuite dissoicée de la polymérase et une troisième étape consistant en la méthylation de guanosine est réalisée par une N7G-méthyltransférase. L’ajout de la coiffeen 5′ au cours de la transcription est associé à uen pause de la machinerietranscriptionnelle. Différentes expériences ont ainsi suggéré qu ecette pause représentait un premier point de contrôle de l’ARN néo-synthétisé car il précède l’étape d’élongation par la polymérase qui n’aurait ainsi lieu que si l’ARN est correctement coiffé. La coiffe est reconnue de façon co-transcriptionnelle dans le noyau par le complexe CBC constitué des protéines Cbp20 et Cbp80 qui lient respectivement la coiffe et le domaine CTD.
Couplage de la transcription et de l’épissage
L’épissage représente le deuxième événement de maturation des ARN pré-messagers. Les gènes eucaryotes sont pour la plupart constitués d’exons (séquences qui se retrouveront dans l’ARNm mature) et d’introns. Bien que la majorité des ARNmde la levure S. cerevisiae ne présentent pas ou peu d’intron, les mécanismes d’épissage ont été relativement bien conservés de la levure à l’homme.
Au cours de la réaction d’épissage, les introns présents dans le transcrit primaire sont excisés et les exons 5′ et 3′ ligués suite à deux étapes de transestérification réalisées par un complexe dynamique, le spliceosome, composé de ribonucléoprotéines: les U snRNP (uridine-riche small nculear ribonculeoproteins) et de nombreux facteurs non-snRNP. Chaque particule U snRNP est constituée d’une molécule U snRNA comexée à sept protéines Sm ou Sm-like ainsi qu’à d’autres protéines spécifiques. Ainsi, la majorité des introns présents dans les ARN pré-messager sont épissés par un spliceosome composé des U snRNP U1, U2, U4, U5, et U6. Les études concernant ce complexe ont été réalisées principalement in vitro et supportent l’hypothèse d’un assemblage dynamique et ordonné de celui-ci sur l’ARN pré-messager. La snRNP U1 s’associe en premier engageant le transcrit vers l’épissage. La snRNP U2 est ensuite ajoutée pour former le pré-splceosome qui est reconnu par un complexe pré-assemblé constitué des snRNP U4, U5 et U6. Différents réarrangements ont lieumenant à la dissociation de U1 et U4 et à la formation d’un complexe d’épissage actif. Cet assemblage est principalement orchestrés 5’SS (splicing site), de point de branchement BP (branching point) et 3′ SS sont très bien conservées parmi les introns présents chez la levure. Ces études in vitro ont été réalisées à partird’ARNm synthétisés préalablement. Or, in vivo, les introns apparaissent à des temps distincts au cours de la transcription. Un assemblage différent du spliceosome in vivo a ainsi été discuté cependant les dernières études réalisées utilisent la technique d’immunoprécipitaion de la chromatine adaptée à l’ARN (CHIP: chromatine immunoprecipitation) tendent à démontrer un assemblage co-transcriptionnel séquentiel comme in vitro.
De plus, certains transcrits sont épissés de façon co-transcriptionnelle. Dans ce cas, on retrouve une implication du domaine CTD de l’ARN polymérase II puisque différentes données exprérimentales supportent l’hypothèse d’un rôle direct du domaine dans la reconnaissance de l’exon (exon recognition). Celui-ci interagirait avec des facteurs d’épissage mais il pourrait également permettre un rapprochement physique des différents exons d’un messager, facilitant ainsi l’assemblage du spliceosome. Il existe un certain nonbmre de différences entre les mécanismes d’épissage chez la levure et l’homme. Il est ainsi intéressant de noter que chez l’homme, un complexe est déposé en amont de la jonction exon/exon après épissage.
Couplage de la transcription et de la maturation de l’ARNm en 3′
La troisième étape de maturation des ARN primaires correspondant à l’ajout d’une queque poly(A) est réalisée de façon concomitante à la réaction de terminaison de la transcription. L’addition de la queque poly(A) est réalisée en deux étapes. L’ARN pré-messager est tout d’abord clivé environ 20 à 30 bases en aval d’un site signal puis est polyadénylé. La maturation d’un ARNm en 3′ peut être reconstituée in vitro avec plusieurs complexes multimériques: CFIA (cleavage factor IA), CFIB (cleavage factor IB) et CPF (cleavage and polyadenylation factor) chez la levure S.cerevisiae. Le premier complexe est constitué de quatre sous-unités, le second d’une protéine alors que le troisième est composé d’au moins quinze facteurs, incluant l’enzyme Pap1 (Poly(A) polymerase) responsable à l’ajout de la queque poly(A) au niveau de l’extrémité clivée 3′-(OH). Les ARNm possèdent une queue poly(A) de taille bien définie puisqu’elle fait en moyenne 70 nucléotides chez lalevure et 250 nucléotides chez l’homme. L’une des questions non élucidées concernant ce processus est de comprendre le mécanisme par lequel la taille de la queue poly(A) est définie. Chez l’homme, il a en effet été établi que l’étape contôlée est la synthèse par un mécanisme permettant une mesure de la taille de la queue poly(A). La réaction de polyadénylation est processive mais une fois la taille de 250 résidus (A) atteint, elle devient distributive et la réaction de polyadénylation est achevée.
Deux hypothèse existent à l »heure actuelle pour la levure. Il a été suggéré que la protéine Pab1 (Poly(A) binding protein) pouvait stimuler la nculéase PAN (poly(A) nuclase), spécifique des résidus (A), pour éliminer l’excès de (A) en digérant l’extrémité 3′. La protéine Pap1 (Pab1 binding protein) qui interagit avec la protéine Pab1 aurait quant à elle un effet négatif sur l’activité du complexe PAN. Cette première hypothèse consisterait ainsi en un mécanisme résultant d’un équilibre entre synthèse et dégradation de la queue poly(A). La deuxième hypothèse implique la protéine Nab2 (Nuclear polyadenylated RNA-binding 2) qui serait responsable d’un contrôle direct de la synthèse de la queue poly(A) comme ceci a été démontré chez les mammifères.
Finalement, il est intéressant de noter que le recrutement des facteurs de clivage est initié dès la reconnaissance du promoteur et que celui-ci est progressif tout le long du gène. Les protéines sont ainsi présentes en forte concentration en 3′ du messager. Ceci peut s’expliquier par différents phénomènes. D’une part, en raison de l’affinité de ces protéinespour l’ARN, on retrouve plus de facterus associés lorsque la taille de cluièci augmente au cours de la synthèse. D’autre part, ces protéines reconnaissent une forme particulière du domaine CTD de la polymérase II qui est phosphorylé au niveau de la sérine 2, modification qui a lieu au cours de l’étape d’élongation. Enfin, il existe une forte spécificité entre les otéines impliquées dans la maturation 3′ de l’ARN pré-messager et le site de polyadénylation. La concentration de ces facterus en 3′ est également ssentielle pour la terminaison de la transcription. Le clivage génère ainsi deux molécules d’ARN. L’ARN possédant la coifffe est polyadénylé alors que l’ARN présentant une extrémité 5′-monophosphate est dégradé rapidement par l’enzyme Rat1. Deux modèles non exclusifs co-existent concernant la terminaison de la transcription. Le premier modèle, appelé <anti-terminateur>, propose que l’apparition des séquences de polyadénylation sur l’ARN en cours de synthèse de maturation de l’extrémité 3′ pourrait éliminer un facteur d’élongation ou bien recruter un facteur de terminaison. L’ARN polyméraseII étant alros moins processive, celle-ci se dissocierait de l’ARN et achèverait ainsi la synthèse du messager. Le deuxième modèle, appelé <toredo>, propose que l’enzyme Rat1 dégraderait l’ARN possédant l’extrémité 5′ mono-phosphate mais ciblerait également la terminaison de la transcription en provoquant la dissaciation de l’ARN polymérase II.
Traduction des ARNm
Au cours de l’initiation, la coiffe recrute la petite sous-unité ribosomale et la grande sous-unité est asemblée au niveau du codon initiateur de l’ARNm avec un ARN de transfert spécifiant une méthionine présent dans sont site peptidyl (P). Durant l’élongation, les ARNt aminoacyls entrent au niveau du site accepteur (A pour aminoccyl) où le décodage prend place. Si l’ARNt est reconnu comme correct, le ribosome catalyse la formation d’un lien peptidique. L’ARNt déacylé et l’ARNt lié à la chaîne peptidique sont ensuite transloqués respectivement vers le site (E) pour (pour exit) et (P) et le codon suivant se retrouveainsi au niveau du site (A). Ce mécanisme est répété jusqu’à l’étape de terminaison qui a lieu lorsque le ribosome rencontre un codon stop. Le peptidefinal est ensuite libéré du ribosome. Les sous-unités ribosomales et les ARNt déacylés sont ensuite dissociés au cours de l’étape de recyclage pour être réutilisés pour un autre cycle de traduction.
Initiation de la traduction:
Assemblage du complexe d’initiation:
La première étape de l’initiation de la traduction correspond à l’assemblage d’un complexe ternaire entre eIF2, une molécule de GTP et un ARNt intiateur (ARNti) spécifiant une méthionine (Met): eIF2-GTP-ARNti_Met. Le facteur eIF2 chargé d’une molécuel de GTP est généré au cours d’une réaction échangeant GDP et GTP catalysée par eIF2B. Cette liaison est stabilisée uniquement quand l’ARNti_Met se lie à eIF2. Après sa formation, le complexe ternaire se lie à la petite sous-unité ribosomale (40S) pour se fixer à l’ARNm à traduire. eIF4E est la protéine cytoplasmique de liaison à la coiffe et l’association de cette protéine à l’ARNm joue un rôle essentiel au cours de l’initiation de la traduction. Elle existe en effet avec la protéine eIF4G au sein du complexe d’initaition eIF4F qui a poru rôle d’ancrer le complexe ternaire au ribosome. Cette liaison est admise comme étant l’étape limitante pour l’initiation de la traduction et est donc soumise à une régulation précise. Nous pouvons ainsi noter que l’initiation des protéine eIF4E et eIF4FG augemente fortement l’affinité d’eIF4E pour la coiffe indiquant certainement leur association avant toue interaction des protéines avec l’ANRm coiffé. La protéine eIF4G joueun rôle central au cours de la liaison de l’ARNm au ribosome. Chez les mammifères, elle est considérée comme une plate-forme adaptatrice puiqu’elle ponte eIF4E et eIF4F respectivement via ses extrémités N et C-terminale. Cette protéine possède en effet plusieurs domaines lui permettant d’interagir avec eIF4E, eIF4F, Pab1 et eIF3 qui est considérée comme une portéine adaptatrice pour le recrutement de l’ARN. En revanche, chez la levure, eIF3 ne lie pas directement la protéine eIF4G mais indirectment grâce à la protéine eIF5. Ainsi, un complexe composé des protéines eIF1, eIF3 et eIF5 permet l’ancrage entre eIF4G et la petite sous-unité ribosomale et ainsi la liaison du complexe ternaire à l’ARNm pour former le complexe 43S. Un complexe constitué des facteurs eIF1, eIF2, eIF3, iIF5 et de l’ARNti-Met a en effet été isolé chez la levure suggérant que la liaison de l’ARNt initiateur à la sous-unité 40S est accomplie suite au pré-assemblage d’un complexe multifactoriel.
L’extrémité 3′ est également essentielle au recrutement de l’ARNm sur la petite sous-unité par l’action conjointe de la queue poly(A) et de la protéine Pab1. La protéine Pab1 se lie le long de la queue poly(A) grâce à quatre domaine RRM (RNA recognition motifs). La structure de la protéine Pab1 a permis de déterminer que le domaine RRM2 contient des sites de liaison pour la protéine eIF4G qui se situent sur la partie opposée à la région liant l’ARN. Cette observation a permis de proposer que cette interaction circularise les ARNm. L’effet de l’interaction entre Pab1 et eIF4F est d’augementer l’efficacité du recrutement du complexe 43S sur l’ARNm. Les effets de la coiffe et de la queue poly(A) ne sont pas additifs mais synergiques in vivo. Ainsi, la queue poly(A) n’est pas suffisante au recrutement du complexe43S. En effet la coiffeest essentielle car elle permet de recruter la petite sous-unité et d’initier la traduction au bon AUG.
Sélection du codon d’initiation AUG:
Le complexe de pré-initiation (48S) recherche par la suite le codon AUG correct. Un mécanisme appelé balayage a été proposé consistant en la progression de la petite sous-unité ribosomale le long de la région 5′-UTR jusqu’au codon initiateur. Ce processus implique la protéine eIF4A qui possède une activité ATPase dépendante de l’ARN capable de dérouler des structures d’ARN in vitro de mnière peu processive. Il s’agit d’un membre de la famille des hélicases à boîte DEAD. L’incorporation d’eIF4A au sein du complexe eIF4F stimule son activité ainsique la protéine eIF4B qui affecte son affinité pour l’ARN et pour l’ATP. Le modèle actuel propose que la protéine peut interagir avec des duplexes d’ARN dans sa conformation liée à l’ATP et que suite à son hydrolyse, elle détruit certains appariements au sein de l’hélice. L’action d’eIF4A est ainsi de dérouler les structures présentes dans la région 5′-UTR assistée de la protéine eIF4B afin de créer un ARN simple brin permettant la liaison du ribosme. Ainsi, le rôle du complexe eIF4F est vraisemblablement de recruter la protéine eIF4A, responsable du déroulement des structures de la région 5′-UTR bien que celle-ci n’appartienne pas directement au complexe eIF4F chez la levure. De plus, nous avons vu qu’il amène le facteur eIF3 qui joue un rôle dans le recrutement du complexe 49S à l’ARNm.
Dest études génétiques ont permis de démonter l’implication des facteurs eIF2, eIF1 et eIF5 dans la reconnaissance du site de démarage AUG et de façon intéressante des liens physiques existent entre ces protéines. Un appariement correct entre le codon AUG et l’anticodon ARNti_Met engendre l’hydrolyse du GTP lié à eIF2 au cours d’une réaction impliquant la protéine eIF5. La protéine eIF5 est une protéine capable d’induire une activité GTPase (GAP: GTPase activation protein) qui est active uniquement lorsque le complexe ternaire est lié au complexe d’initiation. Il a par ailleurs été démontré que la protéine est inactive lorsque le complexe 40S est lié aux factereurs eIF1, eIF1A, eIF3 et eIF5. En revanche, le positionnement ldu complexe au niveau du codon AUG en présence d’eIF5A, eIF4B et d’ATP rstaure l’hydrolyse du GTP par eIF5. Ceci est probablement le résultat d’un remodelage des interactions entre les différents facterus d’initiation suite à un changement de conformation du comolexe ou bien le résultat du départ de facteurs impliquant un rapprochement physique des protéines eIF5 et eIF2.
Suite à l’hydrolyse du GTP, la sous-unité 60S rejoint la petite sous-unité pour former le complexe 80S qui peut alors débuter la formation de lien peptidique entre la methionine et le second acide aminé codé.
Elongation de la traduction:
est une étape très bien conservée contrairement à l’étape d’initiation. Lorsque l’élonation de la traduction débute, un ARNt-aminoacyl se situe dans le site P du ribosme, le site A étant vide. La première étape de l’élongation de la traduction débute avec la formation d’un complexe ternaire entre le facterur d’élongation eEF1A (eukaryotic elongation factor 1A), une molécule de GTP et un ARNt-aminoacyl. Le facteur hétérotrimérique eEF1B catalyse l’échange GDP/GTP sur eEF1A. Ce complexe se lie au site A du ribosme où une vérification s’opère afin de déterminer si l’anticodon de l’ARNt correspond au codon présent au site A. Cette vérification implique de nombreuses étapes telles que l’appariement codon/anticodon, des changements conformaitonnels au sein du cnetre de décodage de la petite sous-unité ribosomale et l’hydrolyse du GTP par eEF1A. Le facteur eEF1A lié au GDP est relargué du ribosome où il laisse l’ARNt-aminoacyl dans le site A qui bascule vers le centre peptidyl transferase du ribosome au cours d’une étape appelé accommodation.
La formation d’un lien peptidique entre l’acide aminé arrivant et l’ARNt portant la chaîne peptidique est ensuite assuré. LARNt ainsi déacylé est déplacé du site P au site E pendant que l’ARNt lié à la chaîne peptidique est déplacé du site A au site P. Cette étape appelée translocation implique la protéine G eEF2 qui hydrolyse alors une molécule de GTP. Le résultat de cette opération est le retour à l’étape initiale avec un ARNt-peptidyl au niveau du site P et le site A vide. L’ensemble du processus décrit est répété jusqu’à ce que le ribosome rencontre un codon stop et initie le processus de terminaison.
Un facteur supplémentaire est nécessaire à la traduction chez la levure et les champignons: le facteur eEF3. eEF3 stimule la liaison de l’ARNt-aminoacyl correct par eEF1A au site A. De plus, il contrôle également l’élimination de l’ARNt déacylé pour faciliter la liaison du complexe ternaire contenant l’ARNt-aminoacyl correct au site A. Il a été montré que eEF3 lie le facteur eEF1A in vitro, cependant son rôle exact au cours de l’élongation n’est pas connu.
Terminaison de la traduction:
Les codons stops rencontrés au cours de l’élongation sont reconnus par un hétérodimère constitué des facteurs eRF1 et eRF3. La cellule ne possède pas en effet d’ARNt capables de reconnaître les codons stops. Les facteurs RF (release factor) de classe 1 à laquelle appartient eRF1 ont été découverts en premier et leur fonction est de trasmettre le singal de la présence d’un codon stop occupant le site A du ribosome au centre peptidyl-transferase. Les troiscodons stops eucaryotes sont reconnus par eRF1 uniquement, contrairement aux procaryotes qui nécessitent deux facteurs. De façon surprenante, les propriété possède plusieurs domaine distincts dont les repliements et dimentsions sont comparables à ceux d’un ARNt. Le domaine N-termianl qui est impliqué dans la liaison au codon stop correspond ainsi au bras de l’anticodon, le domaine central à la tige liant l’acide aminé et le domaine C-terminal à la boucle variable de l’ARNt. Chaque domaine est distinct et conserve ainsi une possibilité de contraintes stériques au sein du site A du ribosome. Un petit domaine a été identifié car celui-ci est très bien conservé. Il s’agit du domaine Gly-Gly-Gln (ou GGQ) dont le rôle est essentiel au cours de la terminaison de la traduction. Les mutants construits ne sont en effet plus capable d’induire l’hydrolyse de l’ARNt-peptidyl alors qu’ils lient toujours le ribosome. Ce domaine n’est pas impliqué dans la reconnaissance du codon stop mais permet vraisemblablement d’interagir avec l’ARt-peptidyl dans le centre peptidyl-transferase du ribosome pour transmettre le signal de la présence du codon stop au centre catalytic et induire l’hydrolyse de l’ARNt-peptidyl.
Une deuxième classe de facteurs RF a été identifiée à laquelle appartient le deuxième facteur de terminaison euaryote: eRF3. Ce facteur possède une activité GTPase qui induit la conversion du GTP en GDP à l’intérieur du ribosome. Contrairement à son homologue procaryote, ce facteur nécessite la présence d’eRF1 pour son activité GTPase et les deux facteurs forment un complexe stable in vivo et in vitro. De façon intéressante, l’interaction est indépendante de l’activité d’eRF1 puisque des mutants de cette protéine sont capables d’activer eRF3. L’activité GTPAse d’eRF3 couplerait la reconnaissance des signaux de terminaison à la libération de la chaîne polypeptidique. L’un des derniers modèles proposés concernant le rôle d’eRF3 au cours de la terminaison est que l’activité GTPase d’eRF3 assiste le facteur eRF1 dans la reconnaissance du codon stop. L’interaction stable entre eRF1 et le site A stimulerait l’hydrolyse du GTP par eRF3. Un changement de conformation pourrait alors s’opérer et permettrrait l’induction de la libération de la chaîne peptidique.
Dégradation des ARNm
Cinq voies de dégradation ont été découvertes chez la levure S. cerevisiae: deux voies dedégradation des ARNm dits <normaux> appelées voies 5′-3′ et 3′-5′ et trois voies de dégradation d’ARNm dits <aberrants> appelées NMD (nonsense mediated deaay), NSD (nonstop decay) et NGD (no-go dacay).
Voie de dégradation 5′-3′:
a été ddéfiniecomme étant la voie majeure de dégradation des ARNm chez la levure S.cerevisiae. Celle-ci peut se diviser en trois grandes étapes. L’ARNm est tout d’abord déandénylé par un complexe constitué des protéines Pop2/Ccr4 jusqu’à ce que la queue poly(A) ne mesure plus que 10 à 15 résidus. Cette étape induit par un mécanisme inconnu le clivage de la coiffe réalisé par le complexe Dcp1/Dcp2 qui libère une extrémité 5′-monophophate, substrat de l’enzyme Xrn1, qui dégrade l’ARNm dans la direction 5′-3′.
Déadénylation:
Un rôle de la queue poly(A) dans la stabilité des ARNm a été proposé très tôt faisant suite à l’observation que les queues poly(A) des ARNm subissent une diminution progressive de leur taille dans le cytoplasme. Il a ainsi été proposé que les queues poly(A) stabilisent les ARNmqui seraient dégradés lorsque celle-ci est diminuée.
Différentes observations ont permis d’établir que la déadénylation était l’étape limitante au cours de la dégradation des ARNm. Ainsi, par des expériences d’induction suivie d’une chasse transcriptionnelle (pulse chase), il a été possbile d’observer la dégradation d’une population homogène d’un ARNm spécifique. Suite à l’arrêt de la transcription de chaque ARN rapporteur testé, un délai variable précédant leur dégradation a pu être mesuré. De façon intéressante, pour les transcrits testés, ce délai est corrélé au temps de leur déadénylation. Cette première observation a suggéré que la déadénylation était une étape requise pour la dégradation des transcrits étudiés et a ainsi démontré son importance. La dégradation de la queue poly(A) n’est pas totale dans un premier temps puisqu’un oligo(A) d’environ 10 à 15 nucléotides demeure à l’issue de cette étape.
Le premier compelxe découvert comme participant à la dégradation de la queue poly(A) chez la levure est le complexe PAN. L’activité de ce complexe est portée par un hétérodimères: Pan2/Pan3. Pan2, la sous-unité catalytique de ce complexe, est un membre de la famille des exonucléases 3′-5′ portant un domaine RNaseD. De façon surprenante, les souches délétées pour le gène PAN2 ou PAN3 ou les 2 sont viable et l’activité déadénylase est epu affectée in vivo, suggérant l’existence d’uatres nucléases responsables de la déadénylation. Identifiée en premier lieu dans des cellules de mammifères, la déadényolase PARN est requise au cours de la maturation des oocytes de xénope et dans des extraits de cellules Hela. Cependant, aucun homologue de cette protéine n’a été découvert chez la levure ou chez la drosophile.
La recherche de nouvelles déadénylases a permis d’identifier le complexe possédant l’activité majeure chez la levure, le complexe Pop2/Ccr4. Différentes observations ont permis sa mise en évidence. Ces protéines appartiennent à deux complexes de 0.9 et 1.9 MDa incluant 5 protéines Not, identifiés comme facteur de transcription car la délétion de chacun de ces gènes engendre une variation des niveaux d’ARNm à l’état stationaire? Une recherche d’homologue du gène humain PARN par alignement de séquences a permis de déterminer que le gène POP2 possède une signature RNaseD tout comme PARN ou PAN2. Ccr4 contient 3 domaines majeurs. Le deuxième domaine est constitué de 5 copies en tandem ‘une séquence répétée riche en leucine (LRR pour leucine-rich repeat) qui est essentiel our son interaction avec la protéine Pop2. Le domaine C-terminal correspond quant à lui à un domaine nucléasede la famille des endonucléases dépendantes du magnésium. De plus, ces deux protéines interagissent avec le facteur Dhh1 impliqué dans la régulation du clivage de la coiffe, étape essentiellem au cours de la dégradation. Les analyses réalisées à l’aide de souches délétées pour le gène POP2 ou CCR4 ont clairment montré l’implication des protéines correspondantes dans la dégradation des ARNm. Les demi-vies d’ARNm rapporteurs stables et instables sont en effet augemntées dans de telles souches. Des expériences d’induction suivie d’une chasse transcriptionnelle ont démontré que ces protéines sont impliquées dans l’étape de déadénylation car la diminution de la queue poly(A) est affectée et imcomplète dans ces souches. Une activité déadénylase résiduelle est cependant présente malgré la délétion de ce complexe. La délétion du gène PAN2 dans une souche délétée pour CCR4 a permis d’établir que cette déadénylation résituelle pouvait être attribuée à l’activité du coplexe PAN. Dans cette souche, les transcrits rapporteurs ne présentent aucune diminution signigicative de la taille de leur queue poly(A) et sont très stables.La viabilité deces soches indique qu’une dégradation des ARNmrésiduelle doit se produire de façon indépendante de la déadénylation. Un défaut d’activité de clivage de la coiffe et de l’activité exolncuoléase 3′-5′ mènent en effet à la mort cellulaire. De plus, l’intermédiaire de dégradation caractéristique de la voie 5′-3′ est présent dans les souches délétées pour POP2, CCR4ou CCR4/PAN2 mais son apparition a lieuplus tard et à un niveau très inférieur à la souche sauvage. Ainsi, ladégradation des ARNm semble se produire de façon indépendante de la déadénylation, activant vraisemblablement un clivage de la coiffe et une dégradation 5′-3′ compte tenu des intermédiareis détectés. Toutes ces observations ont permis de démontrer que le complexe Pop2/Ccr4 était la déadénylase majeure chez S. cerevisiae mais sa mise en évidence a ensuite soulevé la question de la protéine possédant l’activité catalytique in vivo. En effet, à l’issue de ces expéreinces, on peut se demander si ces deux protéines qui interagissent fortemen t possèdent toutes les deux des domaines nucléase actifs in vivo. Deux équopes indépendantes ont finalment mis en évidence qu eCcr4 portait l’activité catalytique dans ce complexe de déadénylation. Afin de déterminer l’importance du domaine nucléase de Ccr4, 4 mutations ont été réalisées parmi les résidus les plus conservés du domaine catalytique putatif. Ces quatre mutants interagissent toujours avec Pop2 mais affectent fortement la physiologie de la cellule qinsi que la stabilité des messagers testés in vivo. La purificatin de la protéine sauvage de levure ainsi que des protéines de souches mutantes a permis de tester leurs activités enzymatiques in vitro. les résultats obtenus ont démontré que la protéines Ccr4 possédait une activité poly(A) exoribonucléase 3′-5’. De plus, la protéine purifiée à partir d’une souche délétée por le gène POP2 présente les m^mes caractéristiques, suggérant que Pop!é n »est pas requise pour son activité. Finalement, la surexpression de la protéine Ccr4 supprime les défauts de croissance et dedéadénylation observés dans une souche délétée pour POP2. De même, la surexpression de Ccr4 dans un contexte sauvage augmente la vitesse de déadénylation des transcrits rapporteurs. Ces mêmes études ont suggéré un rôle des protéines Not associées au complexe Pop2/Ccr4 car les mutations dans les ègnes correspondants engendrenet des défauts de déadénylation. Il est cependant intéressant de noter que l’expressin d’un fragment correspondant au domaine RNaseD de la proteins Pop2 dans E. coli a permis de démontrer l’activité déadényalse de cette protéine, indiquant que Pop2 possède un domaine nucléase fonctionnel. Des analyses de compétitin à l’aide de différents substrats (poly(C), poly(G), poly(A)) a finalmenet établit la préférence de la protéine pour un substrat detype poly(A). Enfin, la structure de la protéine Pop2 a démontré que celle-ci possédait les caractéristiques d’une nucléase. Cependant, il est difficle d’établir son rôle in vivo. Une hypothèse est que la fonction de la protéine est régulée en fonction des conditions de croissnce. En effet, celle-ci est phophorylée maoritairement par le kinase Yak1 en phase stationnaire. Une seconde hypothèse est que la protéine stabilise les interactions du complexe avec l’ARN ou bien que celle-ci est requise pour un adressage correct du substrat à Ccr4.
Voie de dégradation des ARNm aberrants
Il existe ainsi un mécanisme spécifique des ARNm présentant un codon stop précoce, le NMD (nonsense mediated decay), qui permet l’élimination d’ARNm pouvant conduire à la synth_se de protéine tronquées. Il existe également un mécanisme spécifique des ARNm ne présentant pas de codon stop, le NSD (nonstop dacay) qui permet l’élimination d’ARNm pouvant conduire à la synthèse de protéine trp grandes. Finalement, une voie de dégradation (NGD (No-go decay)) des ARNm dont la traduction est bloquée au cours de l’étape d’élongation a été mise en évidence. On comprend aisément qu’il est important pour la cellule d’éliminer ces ARNm aberrants pour d’une part prévenir les effets délétères de ces protéines anormales sur la cellule et d’autre par éviter une utilisation inutile de la machinerie traductionnelle.
Nonsense-mediated decay:NMD
2009-04-26 thèse de Simon Lebaron
La petite sous-unité ribosomique est responsable de l’activité de décodage de l’information génétique contenue dans les ARN messagers. Elles permet l’établissement des interactions entre les codons de l’ARNm et les anticodons des ARN de transfert (ARNr). Elle est donc directement impliquée dans la fidélité de la traduction. La petite sous-unité ribosomqiue est contituée d’une molécule d’ARNr 18S et de 32 protéines chez la levure S. cerevisiae (ARNr 16S et 21 protéines chez E. coli).
La grande sous-unité ribosomique contient le centre peptidyl transférase (PTC) qui catalyse la formation des liaisons peptidiques entre les acides-aminés incoporés. Cette activité est donc nécessaire à l’élongation de la chaîne peptiqdique. La grande sous-unité est composée des ARNr 25/28S, 5.8S et 5S ainsi que de 46 protéines chez les eucaryotes et des ARNr 23S et 5S associés à 34 protéines chez les procaryotes.
l’initiation de la traduction:
débute par la mise en présence de trois éléments: la petite sous-unité ribosomique, l’ANRm et l’ARNt associé à la methionine. Cette étape nécesstie des facterus d’initiation de la traduction: IF chez les procaryotes et eIF chez les eucaryotes. L’association entre la petite sous-unité et l’ARNm, ainsi que la reconnaissance du codon initiateur diffèrent entre les procaryotes et les eucaryotes.
Chez les procaryotes, l’association entre la petite sous-unité et l’ANRm se fait directement via un appariement entre l’ARNr 16S et l’ARNm. Pour cela l’ARNr 16S porte une séquence complémentaire à une séquence nommée Shine-Dalgarno présente sur l’ANRm, à environ 10 nucléotides (nt) en amont du codon initiateur. La petite sous-unité est alors correctement positionnée sur le codon AUG.
Chez les eucaryotes, l’initiation de latraduction débute par l’association de la petite sous-unité au niveau de la coiffe 5′ de l’ARNm. Cette association nécessite l’intervention de facteur eIF mais aussi de la queue polyA et de ses facteurs associés. La petite sous-unité va ensuite scanner l’ANRm en se déplaçant dans le sens 5′-3′ à la recherche du codon initiateur AUG situé dans le bon contexte, c’est-à-dire au sein d’une séquence nommée <Kozak>.
L’ARNt portant la méthionine possède une conformation qui lui permet d’être logé immédiatement au sein du site P, en vis-à-vis du codon AUG, formant ainsi le complexe ternaire dit <d’amorçage>. La grande sous-unité peut alors s’associer avec ce complexe, ce qui correspond à la fin de l’étape d’initiation.
L’élongation de la traduction:
Le processus d’élongation, à la différence de l’initiation de la traduction à été conservé au cours de l’évolution et ne diffère que trèspeu entre les procaryotes et les eucaryotes. Cette étape est composée de plusieurs cycles permettant l’assemblage des acides aminés un à un, chaque cycle comportant trois étapes qui se répètent tout au long du processus d’élongation.
La première étape de ce cycle est le positionnement d’un novuel ARNt amynoacylé au site A. Cette ARNt est apporté par le facteur d’élongation EF-Tu lié à une molécule de GTP. Différentes étapes, mettant en jeu un appariement entre le codon de l’ANRm et l’anticodon de l’ARNt ainsi que différents changements de conformation du centre de décodage de la petite sous-unité, assurent le contrôle de la fidélité de la traduction. Si l’ARNt entrant dans le site A est correct, alors un changement de conformation du facteur EF-Tu intervient, suite à l’hydrolyse du GTP en GDP, ce qui induit une basse de son affinité pour l’ARNt aminacylé et son détachement du ribosome.
La deuxièmeétape consiste en la formation de la liaison peptidique, entre la chaine polypepetidique nassante (au site P) et l’aciede aminé porté par l’ANRt (au site A), par le PTC. Un modèle moléculaire de cette réaction sera présenté ultérieurement.
La troisième étape nécessite l’intervention d’un autre facteur d’élongation, EF-G qui s’associe au ribosome. L’hydrolyse du GTP lié à ce facteur induit la traslocation du ribosome sur l’ARNm. Ce décalage d’un codon exactement (3 nt) libère le site A et positionne l’ANRt portant la chaîne polypepetidique en P et l’ANRt d »acylé dans le site E. Suite à l’hydrolyse de sa molécule de GTP, EF-G se dissocie du ribosome et un nouveau cycle d’élongation peut commencer.
La terminaison de la traduction:
intervient lorsqu’un codon STOP (UAA, UAG ou UGA) au sein de l’ARNm se positionne au nivau du sit eA du ribosome suit à la traslocation de celui-ci. Il n’y a pas d’ARNt ayant un anti-codon complémentaire à ces codon. Les facteurs RF1/2 se positionnent alors dans le site A et induisent l’hydrolyse par le ribosome de la liaison ester unissant la chaîne polypeptidique à l’ARNt dans el site P. Ceci permet la libération du polypeptide. Ensuite, la GTPase RF3 interagit avec les facterus RF1/2 et suite à l’hydrolyse d’une molécule de GTP, ces trois facterus sont éjectés du ribosome.
A la fin de ce processus, une étape de recyclage intervient. Elle permet la dissociation des complexes ribosome/ARNt/ARNm et l’utilisation des sous-unité ribosomiques pour un nouveau cycle de traduction. Ce processus de recyclage nécessite l’intervention de facteurs RRF (ribosome release factor) assistés par le facteur IF3 et EF-G.
le groupe de Noller en 2001 a résolu une structure cristallographique d’un ribosome et en a déduit les structures de la grande et de la petite sous-unité (Yusupov et al.20001).
Chandramouli et ses collaborateurs ont résolu une structure par cryomicroscopie éléctronique d’un ribosome de chien à 8.7A. (Chandramouli et al. 2008).
-
Récent
-
Liens
-
Archives
- Mai 2012 (2)
- décembre 2009 (1)
- octobre 2009 (2)
- septembre 2009 (1)
- juillet 2009 (2)
- avril 2009 (4)
- mars 2009 (6)
-
Catégories
-
RSS
Entries RSS
Comments RSS